|
Inhaltsübersicht | Nanomaschinen | Moleküle | Programme | Kurse | Fun | Links |
||
| > |
Interpretation des genetischen Codes
Der genetische Code wird also dort festgelegt, wo die tRNS Adaptermoleküle mit Aminosäuren beladen werden: In den tRNS-Synthetasen. Für jedes tRNS-Aminosäuren-Paar gibt es eine spezifische Synthetase, die die beiden Moleküle miteinander verknüpft. Die genaue Passform von tRNS, tRNS-Synthetase und Aminosäure bestimmen, welche Aminosäure mit welcher tRNS verknüpft wird. Dabei erkennen viele der tRNS-Synthetasen nicht das Anticodon der tRNS, sondern andere Stellen auf der Oberfläche der tRNS, die mit deren Codon-spezifität nichts zu tun haben.
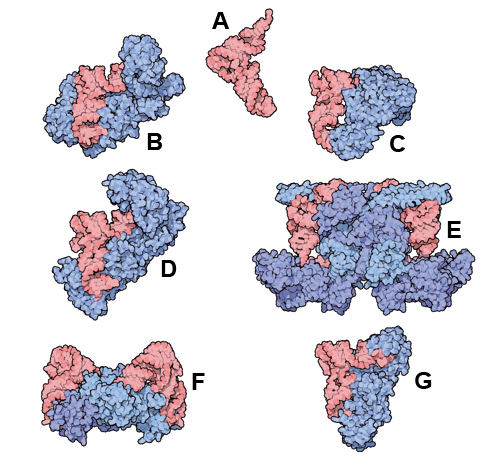
Abb:
A: Typische L-Form einer tRNA; B: Valyl-tRNA-Synthetase; C: Threonyl-tRNA-Synthetase; D: Isoleucyl-tRNA-Synthetase; E: Phenylalanyl-tRNA-Synthetase; F: Aspartyl-tRNA-Synthetase; G: Glutamyl-tRNA-Synthetase.
PDB "Molekül des Monats": tRNA
PDB "Molekül des Monats": Aminoacyl tRNA synthase
Wikipedia: Aminoacyl-tRNA Synthetase, tRNA, Codon, Anticodon