|
Inhaltsübersicht | Nanomaschinen | Moleküle | Programme | Kurse | Fun | Links |
||
| > |
Die digital gesteuerte Proteinsynthese-Maschine
Ribosomen sind Protein/RNS Komplexe, die in großer Zahl im Zytoplasma jeder Zelle vorkommen. Eukaryotische Zellen besitzen zwischen 105 und 107 Ribosomen mit einem Durchmesser von 25 nm. Diese produzieren die Proteine, indem sie das C-terminale Ende eines Peptids von einer transfer-RNS (tRNS) auf die Aminogruppe einer ebenfalls an eine tRNS gebundene Aminosäure übertragen. Dieser Prozess wird Proteinbiosynthese oder Translation genannt (Translation =Uebersetzung, da in diesem Schritt der DNS/RNS Nukleotid-Kode in den Aminosäuren-Kode übersetzt wird) . Die Reihenfolge, in der die Aminosäuren in das wachsende Protein eingebaut werden, ist weder zufällig noch fest in der Struktur des Ribosoms verankert: sie wird bestimmt durch die Basensequenz einer Boten-RNS (mRNS, Messenger-RNS), die die Kopie eines Gens auf der DNS darstellt. Für jede Synthese eines Proteins wird eine neue mRNS geladen. Ein einzelnes Ribosom kann also nacheinander viele verschiedene Proteine produzieren
Die Proteinsynthese wird gestartet durch eine mRNS, die mit Hilfe von Initiationsfaktoren an die kleine Untereinheit des Ribosoms bindet. Nun wird das AUG-Startkodon der mRNS durch die entsprechende tRNS erkannt, die ein Methionin trägt (in Bakterien Formyl-Methionin). Die grosse Untereinheit des Ribosoms kann an diesen Initiationskomplex andocken, die Initiationsfaktoren verlassen den Komplex. In jedem Zyklus der Elongationsphase wird das Peptid um eine Aminosäure verlängert: eine Aminoacyl-tRNS bindet an das nächste freie Nukleotid-Triplett der mRNS, das wachsende Peptid wird auf die Aminosäure an dieser tRNS übertragen, die mRNS wird zusmmen mit dem tRNS-Peptid um ein Triplett verschoben, so dass die nächste Aminoacyl-tRNS an den freigewordenen Platz im Ribosom binden kann. Verschiedene Elongationsfaktoren treiben den Prozess voran und stellen den korrekten Einbau der Aminoacyl-tRNS sicher. Wird ein Stoppsignal auf der mRNS erreicht, so können Terminationsfaktoren binden, das Peptid wird von der tRNS abgepalten und das Ribosom zerfällt in seine Untereinheiten. Diese können ein neues Boten-RNS-Programm laden und mit der Synthese des nächsten Proteins beginnen.
Energieverbrauch: 4 ATP pro eingebauter Aminosäure
Fehlerrate: 1:10’000 Aminosäuren
Geschwindigkeit: 15 Aminosäuren/Sekunde
PDB "Molekül des Monats # 10: Struktur des Ribosoms; # 15: tRNS
Wikipedia: Proteinbiosynthese, Ribosom, Translation, genetischer Code, mRNS, tRNS
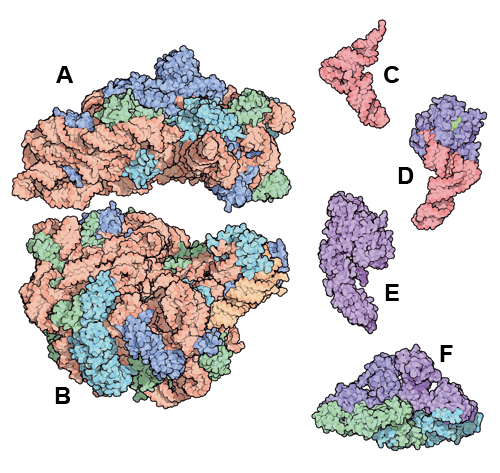 Abb: A: kleine Untereinheit des Ribosoms,, B: grosse Untereinheit des Ribosoms, C: tRNS, D, E, F: verschiedene Elongationsfaktoren
Abb: A: kleine Untereinheit des Ribosoms,, B: grosse Untereinheit des Ribosoms, C: tRNS, D, E, F: verschiedene Elongationsfaktoren
Am Biochemischen Institut benutzt die Forschungsgruppe von Prof. Andreas Plückthun bakterielle Ribosomen, um im Reagenzglas aus Milliarden Genen einer Genbibliothek diejenigen herauszufischen, die ein Protein mit den gewünschten Eigenschaften spezifizieren
weiter: Interpretation des genetischen Codes
zurück: Molekulare Nanomaschinen (Index)