|
Inhaltsübersicht | Nanomaschinen | Moleküle | Programme | Kurse | Fun | Links |
||
| > |
Visitenkarten
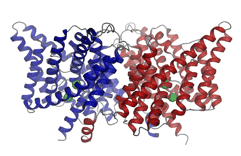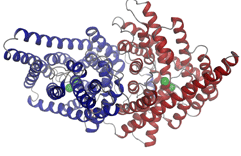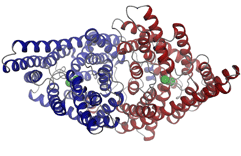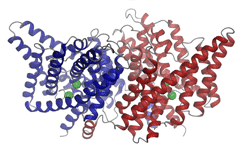 |
Struktur des Chloridkanals.
Proteinkanäle kontrollieren den Transport von Ionen durch die Zellmembran. Die Strukturbestimmung von solchen Proteinen ist besonders schwierig, da sie in der Zellmembran eingebettet sind.Die Arbeitsgruppe von Prof. Raimund Dutzler untersucht die Struktur von Ionenkanälen. |
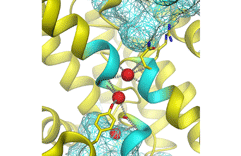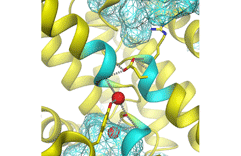 |
Animation der Struktur eines geschlossenen und eines offenen Chloridkanals.
Eine Aminosäureseitenkette bindet im Kanal und verhindert dass die Chloridionen (rot) durch den Kanal fliessen. Im offenen Zustand hat sich die Seitenkette aus der Bindungsstelle im Kanal herausbewegt, die Chloridionen können jetzt durch die Membran fliessen. |
 |
Untersuchung der Faltung einzelner Moleküle mittels Fluoreszenztransfers.
Zwei unterschiedliche Fluoreszenzfarbstoffe werden chemisch an ein Proteinmolekül gekoppelt. Der Farbstoff mit der kürzeren Anregungs-Wellenlänge wird angeregt. Ist das Protein gefaltet, sind die beiden Fluorophore nahe beieinander, die Energie kann vom einen auf den anderen übertragen werden und das System fluoresziert mit der Emmissionswellenlänge des langwelligeren Farbstoffs. Ist das Protein aber entfaltet, ist die Distanz zwischen den Fluorophoren zu gross für die Energieübertragung, und das System fluoresziert mit der Emmissonswellenlänge des kurzwelligeren Fluorophors. In einem Spezialmikroskop kann gezählt werden, wieviele Moleküle mit der kürzeren und wieviele mit der anderen Wellenlänge fluoreszieren. Die Arbeitsgruppe von Prof. Ben Schuler entwickelt Methoden, mit denen sich die Faltung einzelner Proteinmoleküle beobachten lässt. |
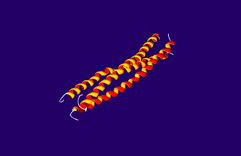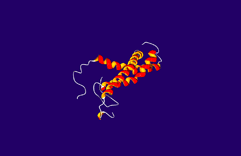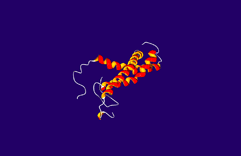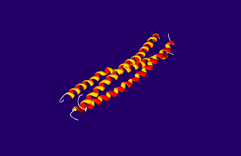 |
Computersimulation der Faltung eines 3-Helix-Bündels
Drei ungefalteten Aminosäureketten (im Bild: grau dargestellt) nehmen eine schraubenförmige Anordnung an (alpha-Helix, im Bild: orange-roter Bereich) und verdrillen sich zu einer Art Seil (coiled coil). Die Arbeitsgruppe von PD Ilian Jelezarov untersucht die Faltzungseigenschaften solcher Helixbündel. Die Funktion der Proteine hängt von ihrer korrekten Falstung ab. Computersimulation helfen uns, zu verstehen, welche Mechanismen dieser Faltung zu Grunde liegen, und wie Mutationen die Faltung und Stabilität eines Proteins verändern. Das Ziel dieser Forschung ist es, eines Tages Algorithmen zu entwickeln, die 3D-Struktur und Stabilität eines komplexen Proteins aus seiner Sequenz vorhersagen zu können. |
 |
Tetramerisierungsdomäne des Tumor-Supressor-Proteins P53
Röntgenkristallographie ist noch immer die wichtigste Methode, wenn es darum geht, die 3D-Struktur von Proteinen zu ermitteln. Dazu muss ein aufgereinigtes Protein dazu gebracht werden, Kristalle zu bilden. Röntgenstrahlen werden an diesen Kristallen gestreut und bilden ein charakteristisches Interferenzmuster, das vermessen wird. Aus diesem Streumuster kann die Anordnung der Moleküle im Kristall und der einzelnen Atome im Molekül berechnet werden. Di Arbeitsgruppe von Prof. Markus Grütter bestimmt auf diese Weise die räumliche Struktur von Proteinen. |
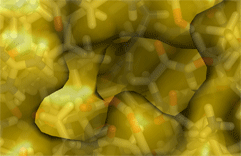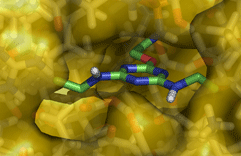 |
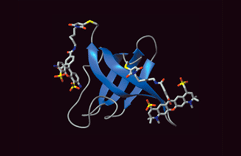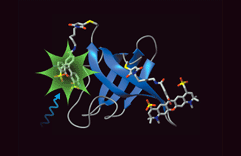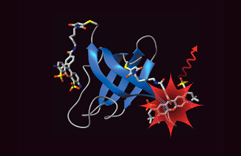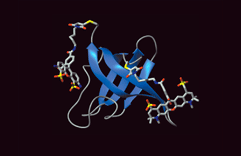
Bindung eines Inhibitors in die aktive Stelle eines Enzyms
Viele Medikamente wirken, indem sie an die aktive Stelle eines bestimmten Enzyms binden und seine Aktivität dadurch hemmen oder unterdrücken. Kennt man die Struktur des Enzyms, kann man mit Hilfe des Computers Moleküle konstruieren, die möglicht gut in die aktive Stelle des Enzyms binden und so den ersten Schritt auf dem weiten Weg zu einem neuen Medikament machen. Die Arbeitsgruppe von Prof. Amadeo Caflisch arbeitet an der Entwicklung von Computerprogrammen, die diesen Schritt automatisieren und die im Computer grosse Substanz-Bibliotheken durchsuchen, um potentielle Inhibitoren eines gegebenen Proteins zu identifizieren. Dies wird dadurch erschwert, dass sowohl die Bindungstaschen in den Enzymen wie auch viele der möglichen Bindungspartner flexibel sind und daher ihre Form verändern können. |
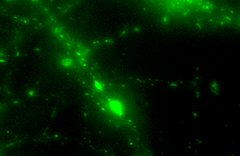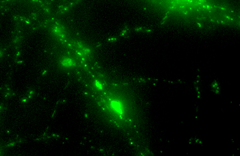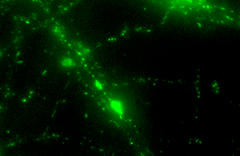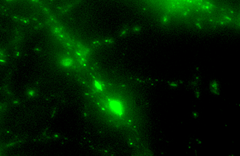 |
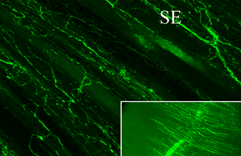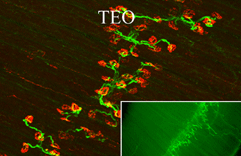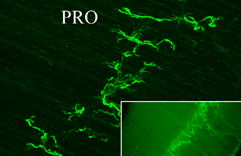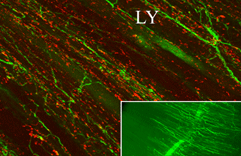
Grün-fluoreszierendes Protein wurde genetisch an die Hirn-Protease Neurotrypsin gekoppelt
Im Fluoreszenzmikrosokop kann man nun sehen, wie mit Neurotrypsin bepackte Vesikel durch die Ausläufer der Nervenzellen zu ihrem Zielort in den Synapsen transportiert werden. Neurotrypsin ist eine Protease, die vor allem in den Synapsen der Nervenzellen zu finden ist. Defekte im Neurotrypsin-Gen können zu schweren geistigen Behinderungen führen. Die Arbeitsgruppe von Prof. Peter Sonderegger entdeckte Neurotrypsin und sein spezifischer Inhibitor Neuroserpin im Zuge ihrer Forschung nach den Mechanismen, mit denen sich die Nervenzellen organisieren |
 |
|